Esta é a página de Citologia Computacional do LAPIX/INCoD/UFSC
Contents
- 1 Diagnóstico Precoce de Câncer na Telemedicina utilizando Citometria Computacional?
- 2 Quais são as potenciais vantagens deste enfoque?
- 3 Nossas Publicações:
- 4 Nossos Datasets:
- 5 Teses e Dissertações
- 6 Método de Coloração #1: Papanicolaou
- 7 Método de Coloração #2: Feulgen
- 8 Método de Coloração #3: AgNOR
- 9 Nossa Ferramenta para Telepatologia/Telecitologia: DIMP-H
- 10 Premiações
- 11 Equipe
- 12 Quer assistir o Webinar do PPGCC?
Diagnóstico Precoce de Câncer na Telemedicina utilizando Citometria Computacional?
Demora no diagnóstico e tratamento do câncer no Sistema ùnico de Saúde é um dos fatores agravantes da doença. Um método simples e que serve para detectar uma ampla variedade de cânceres pode ajudar: a Citologia, que analisa ao microscópio alterações nas células do corpo indicativas de que um câncer pode estar se formando.
Como isso é realizado? Câncer é, a grosso modo, causado por alterações do material genético da célula. Esse material genético encontra-se no núcleo da célula. Então, se analisamos alterações específicas na forma, coloração e tamanho e também nas estruturas do núcleo, podemos identificar um câncer bem antes de, por exemplo, um tumor estar visível. A citologia tem a vantagem de que a coleta da amostra é simples, podendo ser feita por diversos profissionais da saúde treinados e, em alguns casos, pode ser feita com uma simples espátula e/ou escova para fazer uma raspagem superficial.
Outra vantagem é que esse tipo de exame permite detectar o câncer antes mesmo de sintomas aparecerem: você colore as células com um corante que destaca determinadas estruturas das células, fotografa através de um microscópio e a imagem gerada é analisada por um sistema computacional desenvolvido para isso.
Manualmente, ao microscópio, isso é muito laborioso e exige profissionais especializados, mas a Visão Computacional Inteligente baseada em Aprendizado de Máquina Profundo e Redes Neurais Convolucionais pode ajudar. Com uma vantagem: você pode usar isto dentro de um sistema de Telemedicina, como o STT – Sistema Integrado de Telemedicina e Telessaúde que hoje cobre 100% de Santa Catarina e já atende 4 outros Estados. Assim laboratórios do Interior podem corar localmente suas amostras e submeter à análise por um Serviço de Tele-Citologia de uma Rede de Telemedicina, ao invés de enviar amostras ou lâminas por malote para o outro lado do Estado, como ocorre hoje.
É isso que nosso Grupo de Pesquisa em Citometria Computacional aqui na UFSC está pesquisando: Estamos avaliando vários tipos de corantes e diferentes métodos computacionais (veja mais abaixo nesta página) para descobrir qual a melhor forma de realizar o dignóstico da maneira mais precisa e rápida possível. Nós acabamos de publicar alguns resultados interessantes no IEEE Computer-Based Medical Systems 2020e há outraspublicações novíssimas, no forno!!
No nosso grupo estamos focando em duas coisas:
- Melhorar o potencial diagnóstico de exames clássicos de Citologia, como o Papanicolaou: Estamos desenvolvendo métodos de visão copmputacional que objetivam permitir a automatização do processo de análise, a detecção e quantificação de aspectos morfológicos difíceis de reconhecimento ao olho humano e a ampliação do escopo de aplicação, levando este exame clássico para outras áreas além da Ginecologia, como por exemplo a Estomatologia;
- Identificar métodos de coloração com potencial de aplicação à detecção precoce de câncer na Telemedicina e no SUS, como os métodos Feulgen e AgNOR, menos usados, mas com grande potencial. Aqui, por serem exames menos conhecidos e populares, a visão computacional tem o papel adicional de permitir a disseminação desses exames que necessitam de técnicas quantitativas precisas e que poucos especialistas dominam.
Quer saber o que é Tele-Estomatologia? Veja nossa publicação sobre Telemedicina Oral abaixo.
Quais são as potenciais vantagens deste enfoque?
- Eliminar a subjetividade do examinador,
- Poder dar o resultado sem transportar a amostra, implicando maior agilidade no diagnóstico e menor custo para o sistema público de saúde e… mais importante…
- A possibilidade de criar uma tecnologia que detecte o câncer antes do que os olhos humanos iriam fazê-lo, ou seja, dar um diagnóstico ainda mais precoce;
- No futuro, prover um exame que detecte o câncer de maneira simples e precoce e possa ser instituído como uma medida preventiva pública de saúde, por exemplo, como um exame periódico de rotina que pacientes do “grupo de risco pra cancer de boca” poderiam fazer no dentista mesmo, de tempos em tempos. Seriam eles fumantes, etilistas ou que tenham histórico de lesões por HPV em boca.
Nossas Publicações:
- Towards Universal Cytopathology Segmentation: Transformer and CNN Models Across Stains. In: 2025 IEEE 38th International Symposium on ComputerBased Medical Systems (CBMS), 2025, Madrid. 2025 IEEE 38th International Symposium on Computer-Based Medical Systems (CBMS), 2025. p. 343. http://dx.doi.org/10.1109/cbms65348.2025.00077
- Semantic Segmentation for the Detection of Very Small Objects on Cervical Cell Samples Stained with the AgNOR Technique. SSRN Electronic Journal, January 2022. http://dx.doi.org/10.2139/ssrn.4126881
- Computer-Aided Analysis of Oral Conventional Papanicolaou Cytology Samples. SSRN Electronic Journal, January 2022. http://dx.doi.org/10.2139/ssrn.4119212
- Nucleus Detection in Cervical Samples Stained With AgNOR. In: Computer on the Beach, 2022, Itajaí – Santa Catarina – Bras. Anais do XIII Computer on the Beach – COTB’22. Itajaí: Universidade do Vale do Itajaí, 2022. v. 1. p. 045. http://dx.doi.org/10.14210/cotb.v13.p045-050
- What is the State of the Art of Computer Vision-Assisted Cytology? A Systematic Literature Review. COMPUTERIZED MEDICAL IMAGING AND GRAPHICS, p. 101934, 2021. http://dx.doi.org/10.1016/j.compmedimag.2021.101934
- Approaches for the Morphometric Analysis of Feulgen-Stained Cytological Samples. Analytical and Quantitative Cytopathology and Histopathology, v. 43, p. 505-514, 2021
- Segmentation, Detection, and Classification of Cell Nuclei on Oral Cytology Samples Stained with Papanicolaou. SN Computer Science, v. 2, p. 285, 2021. http://dx.doi.org/10.1007/s42979-021-00676-8
- A Novel Approach on Segmentation of AgNOR-Stained Cytology Images Using Deep Learning. IEEE Computer Based Medical Systems 2020. http://dx.doi.org/10.1109/CBMS49503.2020.00110 ;
- Segmentation, Detection and Classification of Cell Nuclei on Oral Cytology Samples Stained with Papanicolaou. IEEE Computer Based Medical Systems 2020. http://dx.doi.org/10.1109/CBMS49503.2020.00018 ;
- Towards a complete pipeline for segmenting nuclei in Feulgen-stained images. Computer on the Beach 2020;
- Systematic Literature Review of Computer Vision-Aided Cytology – A Review of Classic Computer Vision and Deep Learning-Based Approaches published between January/2016 – March/2020. João Gustavo Amorim, Allan Cerentini, Luiz Antonio Buschetto Macarini, André Victória Matias and Aldo von Wangenheim. INCoD/LAPIX.01.2020.E (May, 2020) [PDF] [DOI: 10.13140/RG.2.2.13304.67840]
- Systematic Literature Review of Computer Vision-Aided Cytology – A Review of Classic Computer Vision and Deep Learning-Based Approaches published between January/2016 – March/2020. Relatórios Técnicos do INCoD. http://dx.doi.org/10.13140/RG.2.2.13304.67840 ;
- Localização de Núcleos Celulares em Citologia Oral usando Métodos de Deep Learning. journal of health informatics, v. 12, p. 172-177, 2020.
- Detecção de núcleos em imagens citológicas de AgNOR utilizando Aprendizado Profundo. journal of health informatics, v. 12, p. 178-182, 2020.
- Collaborative Telepathology in a Statewide Telemedicine Environment – First Tests in the Context of the Brazilian Public Healthcare System. IEEE Computer Based Medical Systems 2019. http://dx.doi.org/10.1109/cbms.2019.00139 ;
- Launching a public statewide tele(oral)medicine service in Brazil during COVID-19 pandemic. ORAL DISEASES, v. 2020, p. 1-6, 2020.
- The Use of Neural Network Technology in Automated Grading of Astrocytoma. Pathology, Research and Practice, v. 194, p. 254-254, 1998.
- Data driven approaches for decision making in automated tumour grading. Analytical and Quantitative Cytology and Histology ISSN 0884-6812 , v. 18, p. 298-304, 1996.
- Automated grading of astrocytomas based on histomorphic analysis of Ki-67 and Feulgen stained paraffin sections. Classification results of neuronal networks and discriminant analysis. Analytical Cellular Pathology. ISSN 0884-6812 Analytical and Quantitative Cytology and Histology, v. 8, p. 101-116, 1995
- Computergestützte Graduierung von Hirntumoren. Magazin Forschung der Universität des Saarlandes, Saarbruecken, v. 1994, n.1, p. 30-37, 1994.
- Computer assisted grading of astrocytoma.. Verh. Deutsch. Gesellschaft für Pathologie, v. 78, p. 0/42731-5, 1994.
- Automatic grading of gliomas in stereotactic biopsies. Comparison of the classification results of neuronal networks and discriminant analysis. Clinical Neuropathology, v. 12, p. 253-254, 1993.
Obs.: Algumas publicações acima estão referenciadas por meio do personalized Share Link fornecido pela editora para os autores divulgarem acesso gratuito no momento da divulgação do ahead of print. Pode ser que esse link tenha vencido. Se clicando no link fornecido você não conseguir acessar o artigo, acesse-o pelo DOI.
Nossos Datasets:
Obs.: Estamos preparando uma série de novos datasets, muito mais abrangentes, com mais casos e mais lâminas e com uma curadoria muito mais detalhista. Em breve publicaremo-los! Se você está pensando em desenvolver um projeto nesta área, contate-nos para saber mais.
Teses e Dissertações
Método de Coloração #1: Papanicolaou
A instabilidade cromossômica pode alterar a quantidade de DNA nos núcleos celulares, causando uma condição conhecida como aneuploidia. Considerando que essa alteração parece ter um papel importante no desenvolvimento de câncer, a análise quantitativa de material genético é uma solução promissora e de baixo custo para a detecção precoce de tumores malignos. Apesar disso, essa técnica não é amplamente usada na rotina clínica por conta da falta de um método automatizado para a análise desses dados.
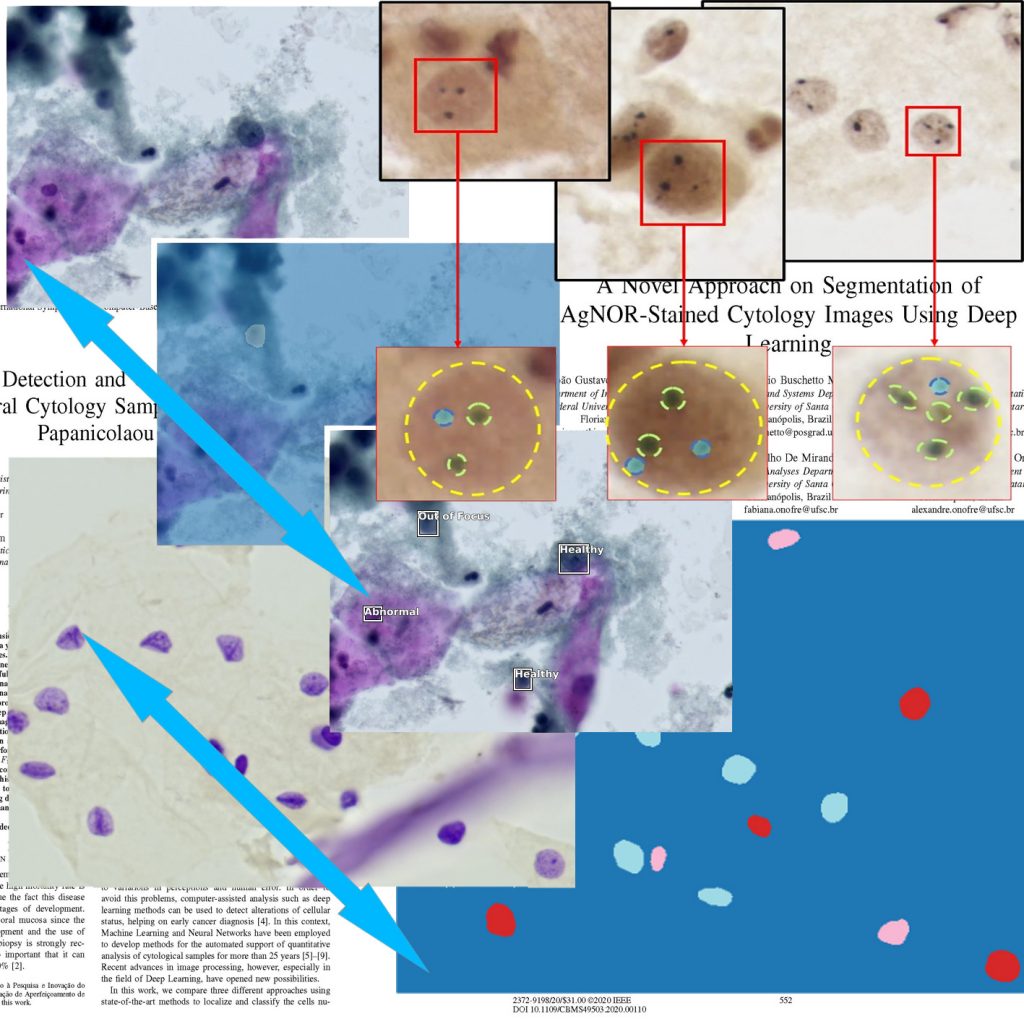
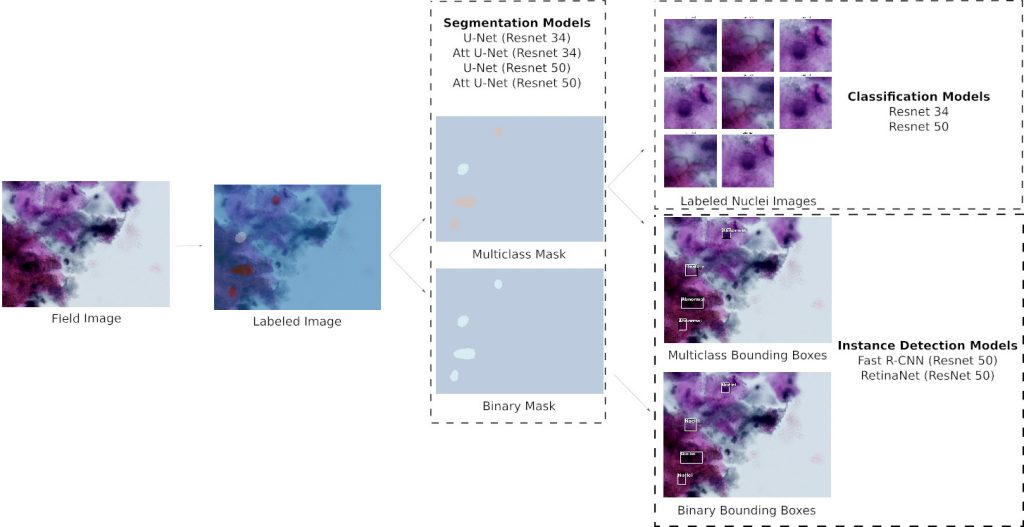
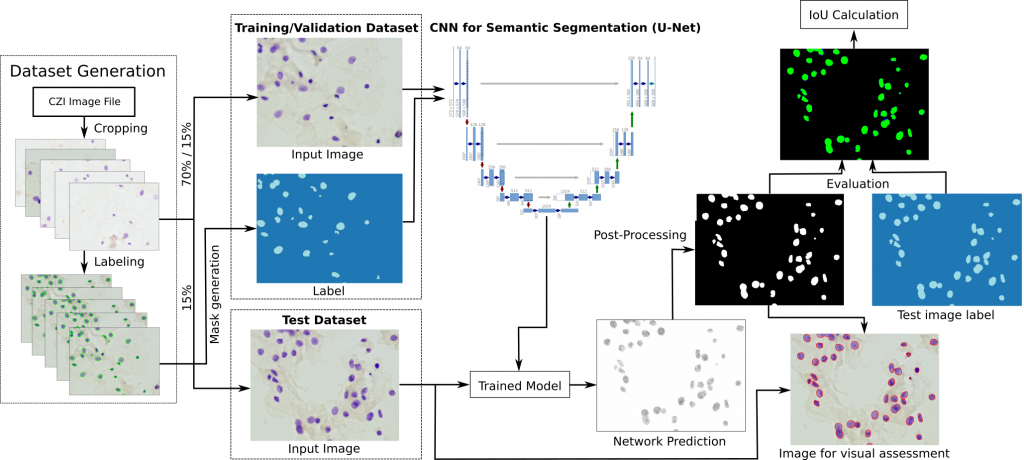
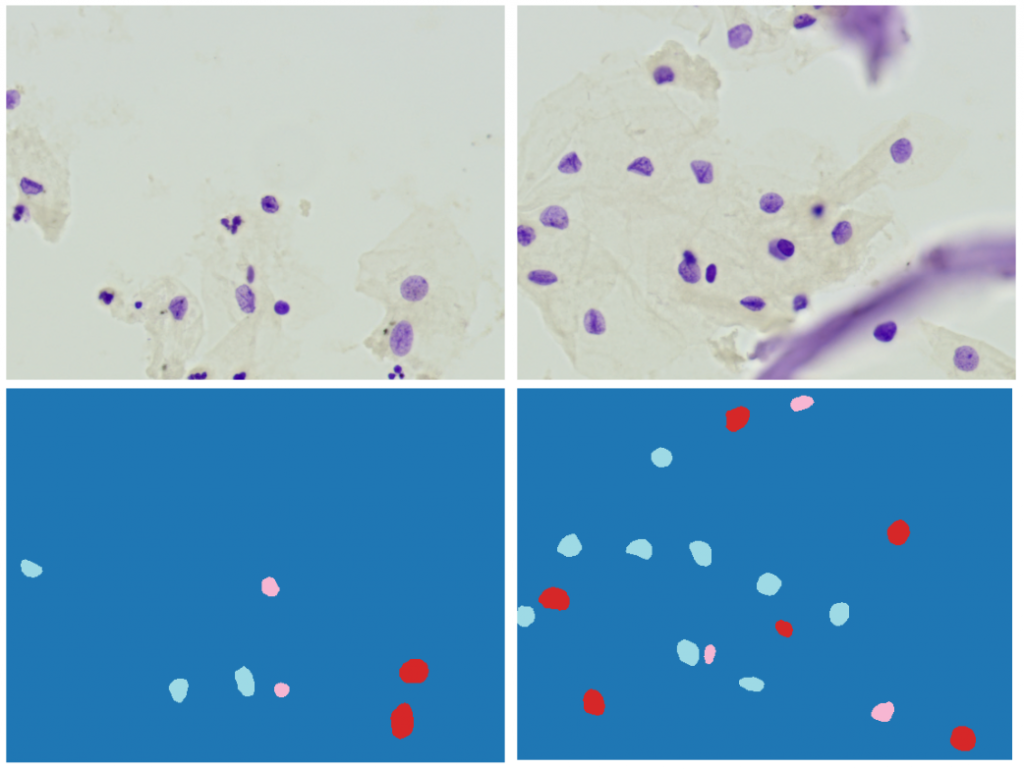
A principal contribuição deste trabalho é a comparação de diferentes abordagens para a automatização da localização, com concomitante classificação, de núcleos na análise de lâminas de citologia oral empregando o tradicional corante de Papanicolau e a técnica de Deep Learning. Papanicolau é uma técnica em uso há mais de 50 anos e bem-dominada mesmo por pequenos laboratórios de análises clínicas do Interior e entendemos que possui, enquanto técnica acessível, alto potencial como porta de entrada para a Tele-Citologia. Neste trabalho foram avaliados modelos empregando duas diferentes abordagens de Deep Learning: segmentação semântica e detecção de objetos. Para a segmentação binária, o modelo U-Net com ResNet 34 como backbone tem até o momento mostrado os melhores resultados, enquanto que, para detecção de objetos, o melhor modelo foi até o momento o Faster R-CNN.
Método de Coloração #2: Feulgen
Mais de 500 mil mulheres são diagnosticadas com câncer de colo de útero todos os anos. Aproximadamente 90% dos casos acontecem em países pobres, mostrando disparidades regionais e globais. Em 2013, o câncer de colo de útero foi a causa da morte de 5430 mulheres no Brasil. O tratamento depende do estágio da doença no momento do diagnóstico e quais são os recursos disponíveis. Este tipo de câncer é curável se detectado cedo e tratado de maneira adequada. Porém, essa detecção precoce ainda é um grande desafio.
Apesar de o método mais comum ainda ser a biópsia, esta detecção precoce pode ser feita utilizando métodos não-invasivos que preservam a função vital do órgão, resultando em uma maior qualidade de vida. A análise morfométrica do conteúdo do núcleo da célula pode ajudar no diagnóstico. Atualmente, a Citometria de DNA é a principal abordagem para uma análise morfométrica visando a detecção e análise da instabilidade cromossômica. Esse processo envolve corar o DNA no núcleo celular de uma amostra de tecido. É geralmente seguido pelo processo de citometria de fluxo ou análise microscópica.
O método mais utilizado para análise quantitativa de DNA em um núcleo celular é a análise de histograma. Parte do processo de análise ainda é feita manualmente por profissionais treinados. Adicionalmente, esse processo de diagnóstico utilizando amostras citológicas consome muito tempo e está sujeita a erros, já que há variações na percepção humana. Para evitar esses problemas, análises por meio de métodos computacionais podem ser utilizadas para detectar alterações no status celular.
O corante de Feulgen é uma reação cito-histoquímica amplamente utilizada, onde a intensidade da coloração é proporcional à concentração de DNA. É mais utilizado para quantificação de DNA em núcleos de células por meio de citometria de imagens, visando fazer a avaliação da ploidia. Alguns autores consideram a coloração de Feulgen mais confiável do que a de Papanicolau, já que a mesma apresenta uma distribuição mais regular de DNA.
Método de Coloração #3: AgNOR
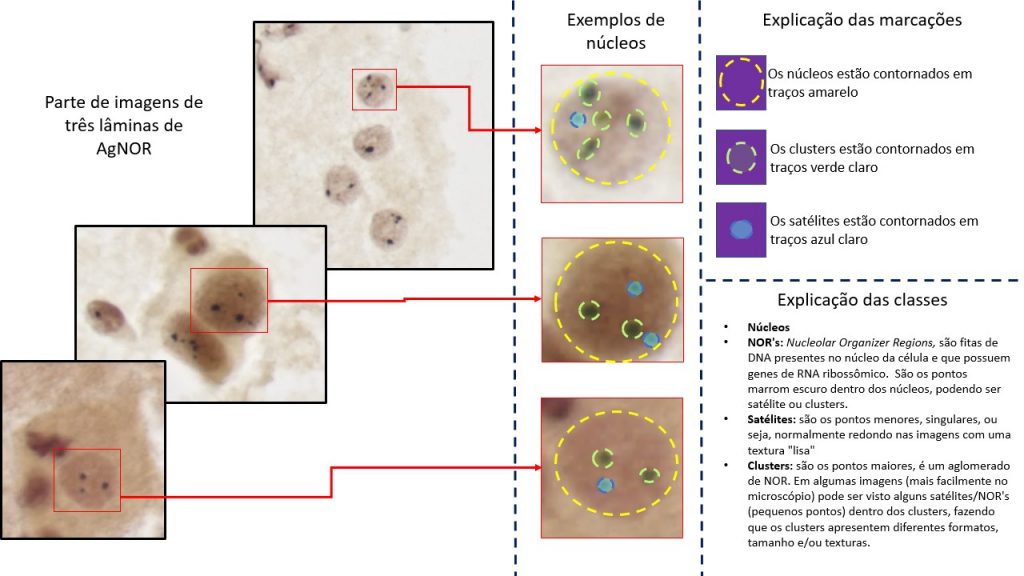
As células cancerosas têm uma quantidade de DNA diferentemaior do habitual, geralmente maior, resultando num processo conhecido como aneuploidia, que está associado à tumorigênese. O aumento da síntese proteica observada nas células aneuploides é um fator que caracteriza a transformação das células de normais para malignas. Desta forma, as Regiões organizadoras nucleolares argirofílicas (AgNORs) podem ser utilizadas como marcador para a quantificação da proliferação celular, diferenciação e transformação maligna(5). O AgNOR tem a vantagem adicional de ser uma técnica simples e barata para detectar a proliferação celular. Vários trabalhos indicam o potencial valor diagnóstico e prognóstico do AgNOR para a citologia cervical. Um destes métodos de diagnóstico é a contagem de NORs, que pode ser facilmente identificada pela técnica de coloração AgNOR.
As Regiões Organizadoras Nucleares (NORs) são loops de DNA nos núcleos de células que têm genes de RNA ribossômico presentes. Estas regiões podem ser identificadas pelo AgNOR porque estão envolvidas por proteínas nucleares que são argirofílicas (prendem-se à prata). Os AgNORs, nos trabalhos realizados até hoje, foram analisados manualmente de acordo com a sua quantidade, tamanho e forma. A analise dos AgNORs utilizando um software requer menos tempo, tem maior precisão e permite a análise de mais células. Esta avaliação manual sofre de vários problemas, incluindo a variação das percepções visuais, uma variação do nível de especialização dos citologistas/patologistas, o tempo de processamento lento da análise manual, entre outros. Estas razões tornam o processo propenso a erros humanos.
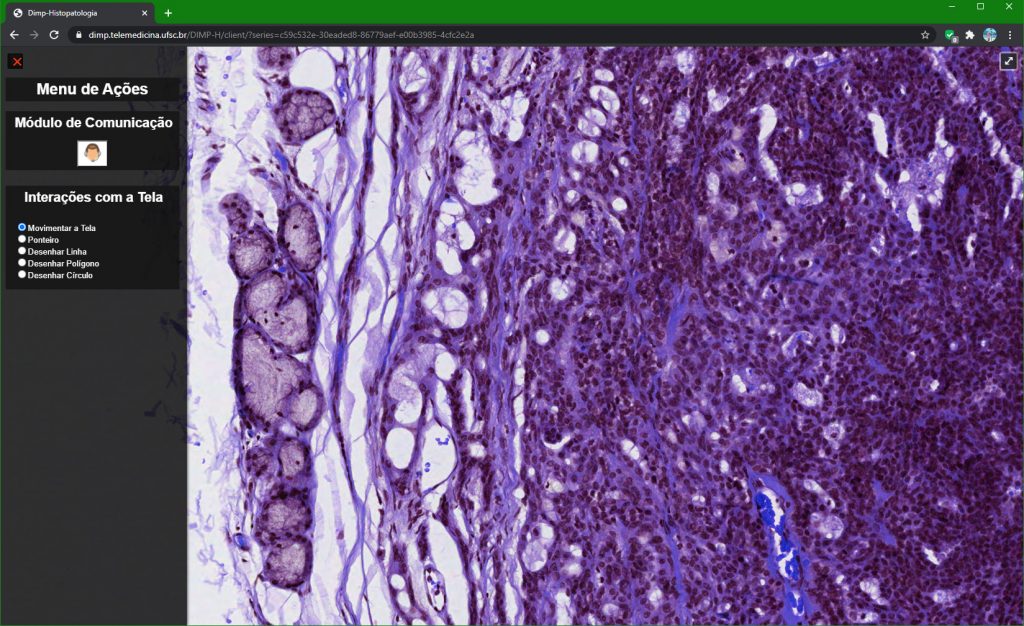
Nossa Ferramenta para Telepatologia/Telecitologia: DIMP-H
O DIMP-H é um visualizador online de lâminas para Telemedicina com Z-stack em formato piramidal DICOM que capacidade de compartilhamento e discussão da lâmina, bem como marcação e desenho, integrado à nossa plataforma de Telemedicina e Telessaúde, o STT.
Veja aqui para saber mais: Collaborative Telepathology in a Statewide Telemedicine Environment – First Tests in the Context of the Brazilian Public Healthcare System. IEEE Computer Based Medical Systems 2019. http://dx.doi.org/10.1109/cbms.2019.00139 ;
Abaixo alguns exemplos de uso com lâminas de Histopatologia e Citologia provenientes de acervos públicos:
Premiações
Melhor Trabalho Completo – Computer on the Beach 2020
Equipe
O Grupo de Pesquisa em Citometria Computacional da UFSC é um grupo de pesquisas multidisciplinar composto por pesquisadores das áreas da Saúde e da Tecnologia, que trabalha de forma conjunta para desenvolver novas metodologias auxiliadas pelo computador para agilizar, disseminar e interiorizar exames médicos de baixo custo mas grande impacto social pela sua capacidade de detectar doenças graves em um estágio inicial.
| Pesquisadores | Filiação |
| Aldo von Wangenheim, Prof. Dr. rer.nat. | Informática e Estatística/UFSC |
| Alexandre Sherlley Casimiro Onofre, Prof. Dr. rer.med. | Análises Clínicas/UFSC |
| Fabiana Botelho de Miranda Onofre, Prof. Dr. rer.med. | Análises Clínicas/UFSC |
| Felipe Perozzo Daltoé, Prof. Dr. | Patologia/UFSC |
| Maria Inês Meurer, Prof . Dr. | Patologia/UFSC |
| Vinicius Andreoli Petrolini, M.Sc. | Telemedicina/UFSC |
| Alunos/Posdocs | Filiação |
| Allan Cerentini, M.Sc. | PPGCC/UFSC |
| André Victória Matias | PPGCC/UFSC |
| Ane Francyne Costa, M.Sc. | PPGFar/UFSC |
| João Gustavo Atkinson Amorim | PPGCC/UFSC |
| Luiz Antonio Buschetto Macarini | PPGEAS/UFSC |
| Analistas de Dados/PETianos & PIBICianos | Filiação |
| Ricardo Thisted Pereira | Eng. Elétrica/UFSC |
| Filipe Landa | Odontologia/UFSC |
| Leonardo Borges Leão | Matematica/UFSC |
| Vinícius Moreno Sanches | Eng. Controle e Automação/UFSC |
| João Henrique Telles | Eng. Mecanica/UFSC |
| Tainee Bottamedi | Farmácia/UFSC |
Quer assistir o Webinar do PPGCC?
Aqui os dados para a sua conexão:
SEMINÁRIOS PPGCC – SEMINÁRIO 04 2020/1
- APRESENTADORES: André Victória Matias, João Gustavo Atkinson Amorim. Mestrandos do PPGCC.
- LINHA DE PESQUISA: Inteligência Computacional
- LABORATÓRIO: LAPIX
- TÍTULO: Uso de Deep Learning para Localização e Classificação em Lâminas de Citologia
- RESUMO: O objetivo do seminário é apresentar os últimos resultados e o andamento da nossa pesquisa sobre a análise de imagens de exames de citologia usando Deep Learning. O exame de citologia é indicado para exames preventivos de câncer e consiste em analisar células em um microscópio de luz. Serão abordados os diferentes tipos de corantes usados nesse exame, o estado da arte da análise computacional desses tipos de dados, as técnicas de segmentação e classificação testadas por nós, as métricas de avaliação utilizadas e os resultados de cada abordagem obtidos até o momento. Por fim, será apresentado o que pretendemos realizar nas próximas etapas para avançar no projeto e superar as dificuldades encontradas.
- DATA: 26/10/2020 (2a feira)
- LOCAL: Plataforma Conferência Web da RNP (https://conferenciaweb.rnp.br/webconf/ronaldo-dos-santos)
- HORÁRIO: 15h30-17h